1) Data 준비
NCBI에서 제공하는 오픈소스 microarray 데이터셋으로 진행하였다(GSE44076). 이 데이터셋은 정상인 50명, 대장암에 걸린 환자 196명의 대장 조직 생검 마이크로 어레이 결과이다. 이 연구에서 GPL13667 microarray chip사용하였으며, 20254개 probe를 통해 암 세포에서 어떤 유전자가 발현되는지 알 수 있다.
2) 알고리즘 설계

NCBI에서 다운받은 데이터는 기본적으로 행 열이 뒤바뀌어 있고 학습에 필요 없는 환자 ID가 존재한다. 따라서 이를 처리해주는 작업을 이전과 같이 진행해 주었다.

변환 결과 모든 실수형 데이터들이 object로 바뀌어 있어 다시 float형으로 바꾸는 작업을 수행하였다.


NCBI에서 해당 데이터의 환자 인덱스와, 일반인 인덱스를 확인하고 레이블링 하는 작업을 수행하였다. 50번째 데이터까지 일반인이고 그 이후로 모두 환자 데이터이다.

레이블 당 데이터의 개수를 확인해 보았다. 환자 데이터 196개, 일반인 데이터 50개이다. 레이블 당 데이터의 개수 차이가 크게 나는 것을 확인할 수 있다. 이럴 경우 데이터가 많은 레이블로 예측할 확률이 높아지기 때문에 오버 샘플링을 필수적으로 수행해야 한다.

학습셋과 테스트셋으로 나눈 후 오버 샘플링을 진행하였다. 기존 학습셋에는 환자데이터 139, 일반인 데이터 33개 존재했고, 오버 샘플링 이후 두 레이블 모두 139로 동일하게 바뀐 것을 확인할 수 있다.

다음으로 랜덤 포레스트 모델을 이용하여 정확도를 테스트해 보았다. 결정 트리 개수는 1000개, 최대 깊이는 100으로 설정하였다. 정확도는 100%가 나왔다. 물론 테스트 셋의 개수가 적은 편이기 때문에 값이 튈 수도 있지만, 이전 실험에 비해 훨씬 높은 정확도가 산출되었기 때문에 어떤 이유로 다음과 같은 결과가 산출된 것인지 확인하기 위해 feature importance 그래프와 변수 histplot을 그려보았다.

Feature importance 그래프는 다음과 같다. 실행할 때 마다 중요 피처의 순위가 계속 바뀌었다. 이는 중요한 피처가 많아 생기는 현상이다. 따라서 각 시행에서 가장 높은 feature importance를 가지는 피처 두 개를 선정하여 그래프로 시각화 해 보았다.

유전자 ‘11743521_s_at’의 환자에서의 발현량과 일반인에서의 발현량을 비교해보았다. x축은 유전자 ‘11743521_s_at’이고 y축은 데이터의 개수이다. 왼쪽이 레이블이 0, 즉 일반인의 발현량 분포이고, 오른쪽이 1, 즉 환자의 발현량 분포 그래프이다. 일반인 발현량 그래프의 경우 약 9.1의 평균값을 가지고, 최소 8.6, 최대9.5 정도의 값을 가지는 정규분포 형태의 그래프이고, 오른쪽은 약 9.7의 평균값을 가지고, 최소 9.3 최대 11.5 정도의 값을 가지는 정규분포 형태의 그래프이다. 평균값이 크게 차이를 보이고, 일반인이 환자에 비해 적은 발현량을 가지는 것을 확인할 수 있다. 또한 일반인의 발현량 최대값이 환자 발현량 최소값과 거의 비슷한 것으로 보아 두 데이터의 분포는 거의 겹치는 부분이 없는 것을 확인할 수 있었다.

다음으로 유전자 ‘11717862_x_at’의 환자에서의 발현량과 일반인에서의 발현량을 비교해보았다. 일반인 데이터는 평균 3.8, 이상치를 제외하면 최소값 3, 최대값 4.6의 값을 가지는 정규분포 형태의 그래프이다. 환자 데이터는 평균 7.2, 최소값 4.6, 최대값 10의 값을 가지는 정규분포 형태의 그래프이다. 앞선 유전자보다 더욱 두 그래프가 겹치는 부분이 존재하지 않는다. 일반인의 해당 유전자 발현량이 훨씬 적은 것을 알 수 있다.
3) 생물학적 분석
위에서 Microarray 결과만으로 중요 feature들을 뽑았다. 그 feature들이 실제로 생물학적으로는 어떤 의미를 가지고 있는지 분석하였다.
- 11743521_s_at : KHSRP, FUBP2 gene의 probe id이다. KHSRP(KH-Type Splicing Regulatory Protein)는 단백질 코딩 유전자이다. KHSRP와 관련된 질병에는 척추 근육 위축 이 포함된다. KHSRP 유전자는 전사, 대체 pre-mRNA 스플라이싱 및 mRNA 국재화를 포함한 다양한 세포 과정에 관련된 다기능 RNA 결합 단백질을 인코딩하는 역할을 한다. 또한 종양 생성과 전이를 유도한다.
- 11717862_x_at : EGR1, KROX24, ZNF225 gene의 probe id이다. 이들은 재조합단백질이다. EGR1은 핵 단백질이며 전사 조절자 역할을 한다. EGR-1이 활성화하는 표적 유전자의 산물은 분화 및 유사분열에 필요하다. 연구에 따르면 이것이 종양 억제 유전자라고 한다. EGR-1은 뇌에서 뚜렷한 발현 패턴을 가지고 있으며, 그 유도는 신경 활동과 관련이 있는 것으로 나타났다.
- 11749905_a_at: CYR61, CCN1, GIG1, IGFBP10 gene의 probe id이다. 주로 지방조직에서 발현한다. 세포 증식, 화학주성, 혈관형성 및 세포 부착을 촉진한다. 이 유전자에 의해 코딩되는 분비된 단백질은 성장 인자-유도성이고 내피 세포의 부착을 촉진한다. 단백질은 또한 세포 증식, 분화, 혈관신생, 아폽토시스 및 세포외 매트릭스 형성에 있어서도 역할을 한다.
- 11754334_s_at: EGR1, KROX24, ZNF225 gene의 probe id이다
- 11743444_s_at:RHOB, ARH6, ARHB gene의 probe id이다. 이들은 주로 ECM조직에서 발현된다. DNA 손상 후 신생물로 형질전환된 세포에서 아폽토시스를 매개한다. 발달에 필수적이지는 않지만 형질전환 된 세포에서 세포 부착 및 성장 인자 신호 전달에 영향을 미친다. 결실이 종양 형성을 일으키기 때문에 종양 발생에 부정적인 역할을 한다.
- 11715354_a_at: COL1A2 gene의 probe id이다. 단백질 코딩 유전자이다. COL1A2와 관련된 질환은 골형성 불완전, 유형 iii 및 골형성 불완전성, 유형 IV를 포함한다. 관련 경로 중에는 ERK의 Integrin Pathway 및 Development Angiotensin 활성화가 있다.
- 11752940_a_at: EGR1, KROX24, ZNF225 gene의 probe id이다.
암은 세포주기를 조절하지 못하고 세포 증식을 지속적으로 하는 질병이다. 따라서 암에 걸린 환자들의 세포에서는 세포증식을 유도하는 유전자(oncogene)는 많이 발현되고, 세포 주기를 조절하는 유전자들(tumor suppressor)은 발현이 적을 것이다. 중요 feature들 중에서 EGR-1, CYR61, ARH6, KHSRP 등은 세포 주기와 관련 있는 인자들이다. 또한 암이 증식하기 위해서 많은 양분이 필요하므로 혈관 형성을 유도하는 인자들을 분비한다. COL1A2과 CYR61는 혈관 신생을 유도하는 역할을 한다. 따라서 암과 관련된 인자들이 중요 feature로 뽑혔으며, 우리가 구축한 모델이 생물학적으로도 유의미하다.
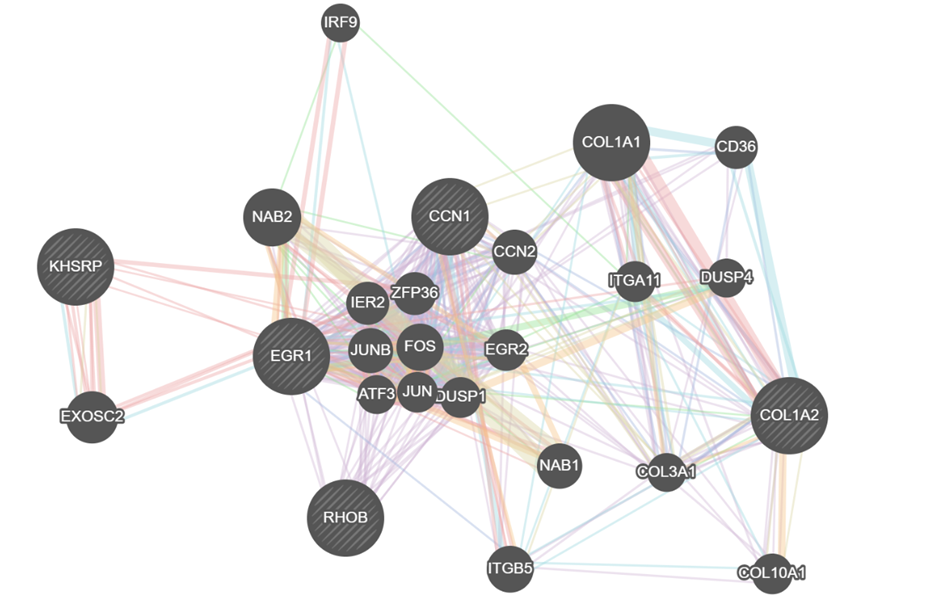
중요 feature들 사이의 연관성을 보기위해서 geneMANIA database를 이용하여 gene network를 그렸다. 이들은 밀접한 연관이 있으며, MAPK pathway와 관련된 유전자들과 연관이 많이 되어 있다. 즉 세포 주기를 조절하는 인자들과 밀접한 연관이 있다.
'Project > 드림학기제_Machine Learning 암 발병률 예측' 카테고리의 다른 글
| 대장암 혈액 진단 (0) | 2022.07.11 |
|---|---|
| 신장 질환 진단 모델 (0) | 2022.07.11 |
| 전립선암 진단 모델 (0) | 2022.07.11 |
| 프로젝트 개요 및 이론적 배경 (0) | 2022.07.11 |
| 1. 드림학기제_암 발병률 예측 프로그램(with 생물정보학) Intro (0) | 2022.05.09 |